주로 단백질-단백질 상호 작용 (protein-protein interaction, PPI)나 gene-gene interaction에 의한 network을 분석할 필요가 있을 때, 또는 microarray 결과를 이용하여 differentially expressed gene (DEG) 를 찾고, 그것으로부터 Gene Set Enrichment Analysis (GSEA)나 EASE 로 변화된 signal pathway (주로 '모듈'로 표현되는) 을 찾아 시각화 하고자 할 때 사용할 수 있는 프로그램, Cytoscape. 간단히 PPI를 예로 들면, 엑셀 파일을 불러 올 수 있기 때문에 사용하기 편하고, simple interaction format 이란 sif 역시 만들기 쉽다. 또한 노드나 에지의 속성을 변화시켜서 어떻게 시각화할 것인가를 결정하는 vizmapper (visual mapper) 역시 만들고/수정 하기 쉽게 되어 있다.
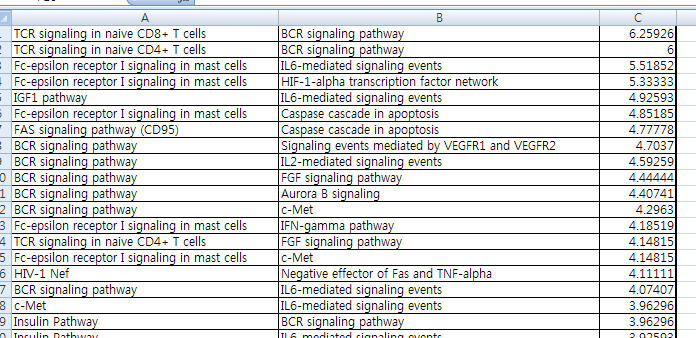
이렇게 되어 있는 엑셀 파일을 불러 오기 위하여 메뉴에서 "File --> Import --> Network from Table (Text/MS Excel)..." 을 선택하면 다음과 같은 대화 상자가 나타난다.
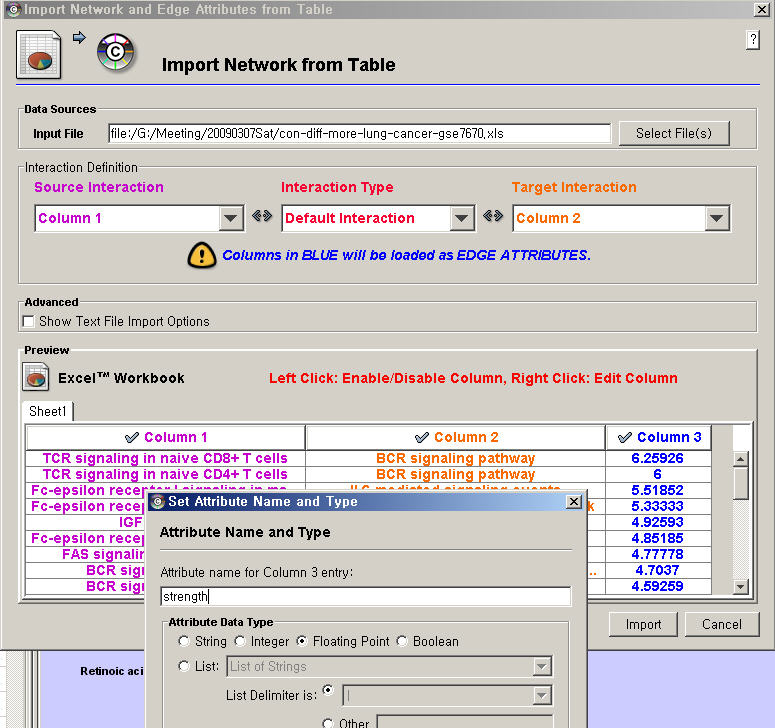
network 파일을 import하는 화면.
첫 번째 컬럼과 두, 세 번째 컬럼을 import 할 것이므로 "Preview" 윈도우에서 그 컬럼들을 마우스 왼쪽 클릭해준다. 그 후 위의 "Interaction Definition"에서 source interaction 으로 쓰일 컬럼을 선택한다. 그리고 "Target Interaction"으로 쓰일 것도. 그리고 Interaction Type 을 설정할 수 있는데, 그냥 설정된 기본값을 사용한다. 엑셀 파일의 첫 번째 컬럼과 두 번째 컬럼이 서로 interaction 하는 노드를 차례로 적어 놓은 것이라고 할 때 위처럼 설정하면 된다. 또한 세 번째 컬럼이 interaction 의 강도 였다면, 아래 "Preview" 화면에서 "Column 3"을 마우스 오른쪽 클릭하면 "Set Attribute Name and Type" 이라는 작은 대화 상자가 또 뜨는데, 그 곳에 적당한 값을 설정해 주면 된다.
그리고 Import 를 시키고 layout 적당히 해주고, edge 두께를 strength 에 맞게 해주면 다음과 같은 network 을 간단히 그릴 수 있다.
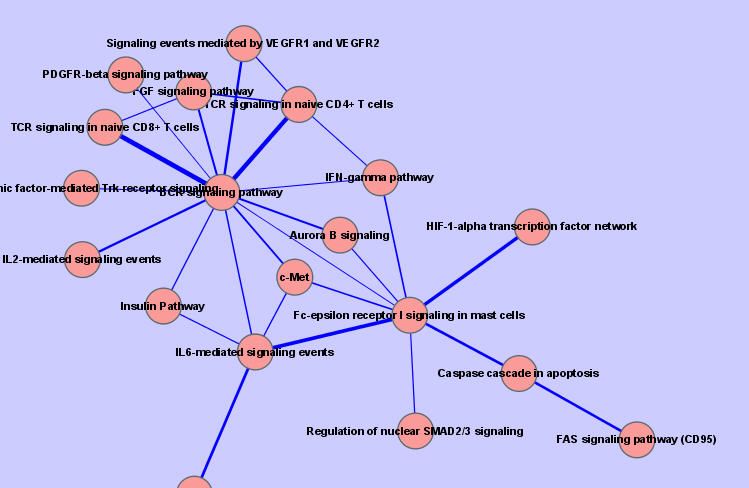
cytoscape으로 그린 network의 예
이 프로그램, 정말 잘 만들었다. 예전에도 느꼈지만. 게다가 버전 조금 올라가면서 매우 많이 바뀌었군. 특히 VizMapper. delphi처럼 attribute 를 바꾸기 쉽게 해 놓았어... 이런저런 control 써서 UI 편하게 한 것이야 뭐 약간의 성의가 있으면 된다 하지만, passthrough mapper 설정하는 UI, 정말 끝내주는군, 이런 건 단순히 컨트롤 갖고 끄적된다고 될 것이 아니라, 정말 창의적으로 처리했다는 것을 알 수 있다.
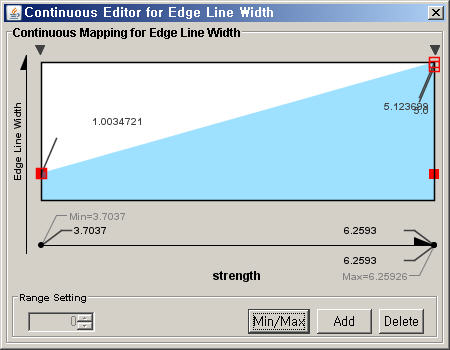
continuous mapper의 설정 화면. 최대/최소값을 중심으로 값을 mapping 시킬 범위를 설정하는 화면.
strength 에 따라 edge의 두께를 다르게 하고 싶다, 라면 strength 가 x 축, edge 두께가 y 축이 되어 어떤 식으로 mapping 할 것인지를 저 red open square를 움직여서 설정하게 되어 있다. 완전 감동.
포토샵과 비지오 이후 오랜만에 프로그램 자체에 놀라게 되는구나. 언제쯤 이런 멋진 프로그램을 만들 수 있을까?
'연구관련 > Bioinfo류' 카테고리의 다른 글
| 정규화(normalization) (18) | 2010.04.19 |
|---|---|
| drug 관련 싸이트 (0) | 2010.04.06 |
| 생물정보학(bioinformatics)에 대하여 (3) | 2010.01.12 |
| outlier 빼고 상관계수 구하기 : Mahalanobis 거리 (1) | 2010.01.04 |
| ROC의 AUC 구하기 (17) | 2010.01.04 |
