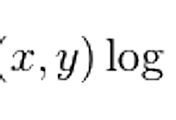Connectivity Map (CMap) 은 1,308 개의 chemical 을 몇 가지 cell-line 에 treatment 하기 전/후에서의 gene expression profile 을 microarray 로 측정한 결과를 제공해 주는 database 이다. raw CEL file 과 각 실험 조건에 관한 정보 파일을 download 할 수 있기는 한데, 문제는 각 실험에 사용된 chemical 의 정보가 이름밖에 없다는 것이다. 보통 chemical 이름은 동의어가 여럿 있기 때문에 이 이름만 갖고 일을 하는 것은 쉽지 않다. CCLE나 DrugBank, TTD, GDSC, PubChem, TCM@Taiwan, Chembl, clinicaltrails.org 에 있는 자료랑 같이 사용할 때 동일한 chemical 에 대해서 각 DB마다 서로 다른 이름을 사용할 수 있기 때문에, 각 chemical 의 정보를 최대한 많이 가져 와서 제대로 matching 시키는 일이 필요하다. 보통 다른 DB 같은 경우 자신의 DB에 사용된 chemical 에 대한 PubChem 을 비롯한 다른 DB에서 사용된 그 chemical 의 ID 를 제공해 줘서 이런 일을 쉽게 할 수 있는데, CMap 은 그렇지 않아서 약간 돌아 가야 한다. 그 방법을 살펴 본다.
CMap 에서 instance_id 는 기본적으로 chemical 에 대한 primary key 이다. 즉, 동일한 primary key 를 갖는 chemical 은 같다. 그런데 instance_id 는 그 ID 만 있을 뿐 다른 정보가 없다. 대신, 정보가 ChemBank 에 저장이 되어 있는데, 문제는 ChemBank 에서 사용하는 cbid 와 instance_id 가 일치하지 않으며, 그 전환 방식 또한 밖으로 드러 나지 않는다는 것이다. ChemBank 에 PubChem ID 가 있기 때문에 instance_id of CMap 과 cbid of ChemBank 만 알면 쉽게 할 수 있는데 그렇지 않으니 이 부분을 직접 건드려야 한다.
우선 ChemBank 에서 chemical provider 로 검색을 한다. CMap 에는 총 26개의 화학 시약 회사의 제품이 사용되었고, 이 정도면 직접 손으로 검색해 볼 만 하다. 각 provider 로 검색한 결과를 ChemBank 에서 text file 로 export 할 수 있고, 그 file 안에는 ChemBank ID 와 SMILES 가 있다. SMILES 로 비교하는 것도 완벽하게 되지 않기 때문에 일단 ChemBank ID 를 갖고 일을 더 진행해 보자. ChemBank 에서는
http://chembank.broadinstitute.org/chemistry/viewMolecule.htm?cbid=3187756
형식으로 cbid 를 이용하여 그 chemical 의 정보 페이지를 download 할 수 있다. 이런 식으로 각 chemical 의 정보 페이지를 download 한 이후, 각 정보 페이지에 나와 있는 이름과 CMap 에 있는 이름을 비교하여 CMap 의 instance_id 와 ChemBank 의 ID 를 mapping 시킨다. 그 후 ChemBank 에서 download 한 각 chemical 의 정보 파일에 있는 PubChem ID 등을 사용하여 CMap 에 사용된 chemical 의 여러 정보를 얻어 올 수 있게 된다.
이 때, 물론 하나의 instance_id 에 해당하는 이름이 여러 ChemBank ID 의 이름과 일치할 경우, CMap에서 제공하는 provider 정보, 즉 그 chemical 을 판 회사 이름과 그 회사 제품 번호를 이용하여 chemical 의 정보를 일일이 검색해서 ChemBank 의 여러 ID 중 어느 것이 맞는지를 일일이 확인해 줘야 한다.
저렇게 웹페이지 수백, 수천개를 짧은 시간 안에 전부 받아 오는 것은 서버 쪽에서 막을 수도 있다. 지금 ChemBank 서버가 그것을 막아 놓았는지 확인해보진 않았지만 어쨌든 너무 자주 사용하는 것은 좋지 않다. 예전에 학생 한 명이 Nature 였는지 Science 였는지에서 논문 수백개를 저런 식으로 download 해서 kaist 전체가 그 논문 site 접속이 아예 막혔던 적이 있다, >.<""
URL 을 이용하여 web-page 를 download 하는 방법은 난 linux 의 wget 을 사용하는데, python 의 urlretrieve 나 MS-Windows 에서는 URLDownloadToFile API 를 사용하면 쉽게 할 수 있다.
SMILES나 InChI를 이용해서 비교를 해도 잘 안 되는 경우가 종종 있다. 즉, 동일한 chemical 에 대한 이 값들이 다른 경우가 종종 있다. 그래서 내가 했던 방법은 mol 이나 sdf 파일을 이용해서 구조적으로 비교를 하는 것이었는데 이것은 시간이 좀 오래 걸리긴 한다. 어쨌든 동일한 chemical 인지를 판단하는 일은 쉽지 않기 때문에 저렇게 cross-matching ID 를 이용하는 방법이 그나마 괜찮다, 각 DB 자체에서 제공하는 정보는 믿을 수 있다는 가정 하에.
'연구관련 > Bioinfo류' 카테고리의 다른 글
| Mutual Information 추정하기 (0) | 2014.04.13 |
|---|---|
| pathway database 종류 및 특성 (0) | 2014.03.18 |
| R의 Bioconductor 및 관련 package 를 설치하면서 (0) | 2013.11.22 |
| 기계학습 관련 글 예고(?), ㅋ (0) | 2013.09.29 |
| chemical database 인 Chembl DB의 ERD (0) | 2012.07.23 |